8. Análisis de Componentes Principales#
8.1. Planteamiento1#
Se aplica a tablas de datos donde las filas son considerados como individuos y las columnas como datos cuantitativos.
Más formalmente, se dispone de los valores de \(p\) variables y \(n\) elementos dispuestos en una matriz \(\mathbf{X}\) de dimensión \(n\times p\).
Siempre (casi) se usa la matriz centrada y/o estandarizada, los paquetes suelen hacer este trabajo por nosostros. Supongamos que \(\mathbf{X}\) ha sido centrada, su matriz de varianza covarianza viene dada por \(\frac{1}{n}\mathbf{X'X}\).
¿Cómo encontrar un espacio de dimensión más reducida que represente adeucadamente los datos?
8.1.1. Notación#
Se desea encontrar un subespacio de dimensión menor que \(p\) tal que al proyectar sobre él los puntos conserven su estructura con la menor distorsión posible.
Consideremos primero un subespacio de dimensión uno (una recta) obtenida por un conjunto de \(p=2\) variables.
La siguiente figura indica el diagrama de dispersión y una recta que, intuitivamente, proporciona un buen resumen de los datos, ya que las proyecciones de los puntos sobre ella indican aproximadamente la situación de los puntos en el plano.
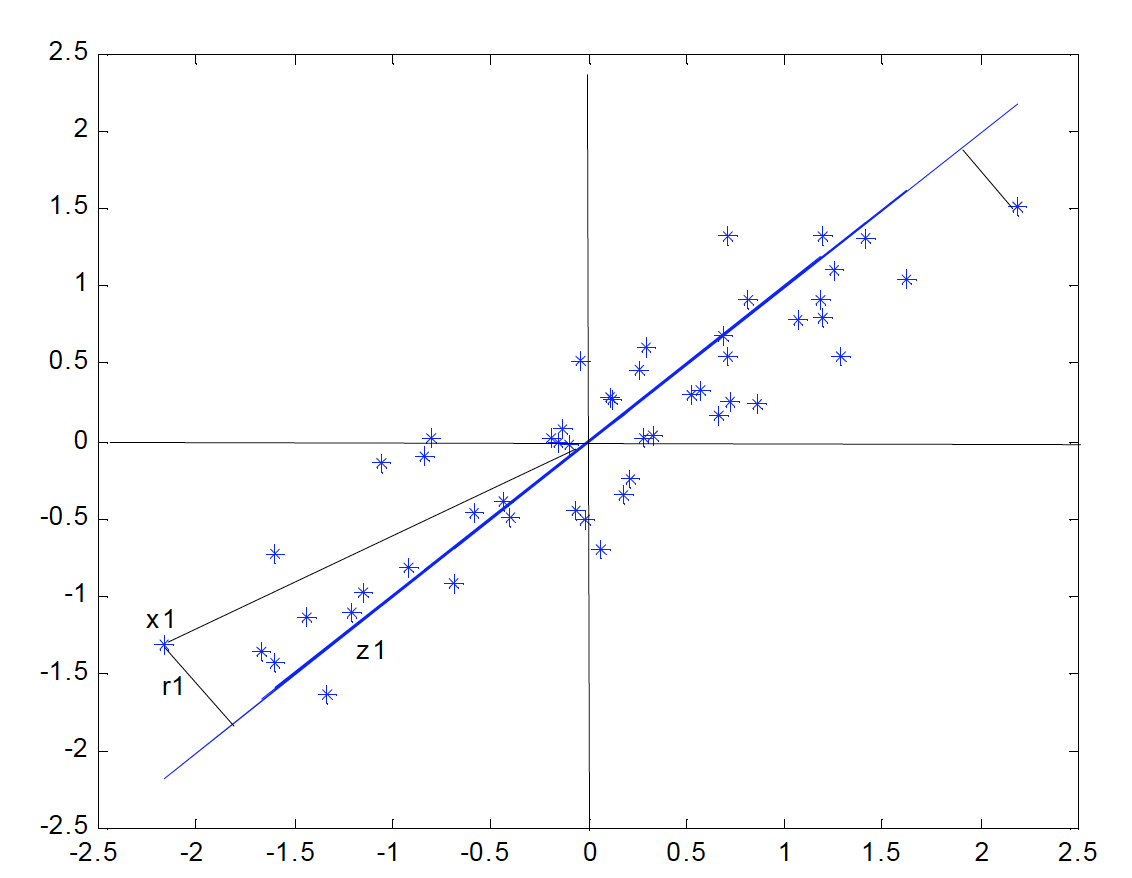
Si consideramos un punto \(\mathbf{x_i}\) y una dirección \(\mathbf{a_1} = (a_{11}, \ldots, a_{1p})'\), definida por un vector \(\mathbf{a_1}\) de norma unidad, la proyección del punto \(\mathbf{x_i}\) sobre esta dirección es el escalar:
y el vector que representa esta proyección será \(z_i\mathbf{a_1}\). Llamando \(r_i\) a la distancia entre el punto \(x_i\), y su proyección sobre la dirección \(\mathbf{a_1}\), este criterio implica:
donde \(|\cdot|\) es la norma euclideana o módulo del vector.
Notemos que al proyectar cada punto sobre la recta se forma un triángulo rectángulo donde la hipotenusa es la distancia al origen del punto al origen, \((\mathbf{x_i'x_i})^{1/2}\), y los catetos la proyeccion del punto sobre la recta \((z_i)\) y la distancia entre el punto y su proyección \((r_i)\). Por el teorema de Pitágoras, podemos escribir:
y sumando esta expresión para todos los puntos, se obtiene:
Como el primer miembro es constante, minimizar \(\sum_{i=1}^{n}r_i^2\), la suma de las distancias a la recta de todos los puntos, es equivalente a maximizar \(\sum_{i=1}^{n}z_i^2\), la suma al cuadrado de los valores de las proyecciones. Como las proyecciones \(z_i\) son variables de media cero, maximizar la suma de sus cuadrados equivale a mazimizar su varianza.
¿Cómo es eso posible?
8.1.2. Cálculo del primer componente#
El primer componente principal será la combinación lineal de las variables originales que tenga varianza máxima. Los valores de este primer componente en los \(n\) individuos se representarán por un vector \(\mathbf{z_1}\), dado por
Como las variables originales tienen media cero también \(\mathbf{z_1}\) tendrá media nula. Su varianza será:
donde \(S\) es la matriz de varianzas y covarianzas de las observaciones. Para que la maximización de la ecuación anterior tenga solución debemos imponer una restricción al módulo del vector \(\mathbf{a_1}\), y, sin pérdida de generalidad, impondremos que \(\mathbf{a_1^{'}a_1}=1\). Usamos para ello el multiplicador de Lagrange
Se maximiza derivando respecto a los componentes de \(\mathbf{a_1}\) e igualando a cero. Entonces
cuya solución es:
que implica que \(\mathbf{a1}\) es un vector propio de la matriz \(\mathbf{S}\), y \(\lambda\) su correspondiente valor propio. Para determinar qué valor propio de \(\mathbf{S}\) es la solución de la ecuación tendremos en cuenta que, multiplicando por la izquierda por \(\mathbf{a'_{1}}\) esta ecuación,
y concluimos, que \(\lambda\) es la varianza de \(\mathbf{z_1}\). Como esta es la cantidad que queremos maximizar, \(\lambda\) será el mayor valor propio de la matriz \(\mathbf{S}\). Su vector asociado, \(\mathbf{a1}\), define los coeficientes de cada variable en el primer componente principal.
Ejemplo
El siguiente conjunto de datos corresponde a calificaciones de 20 estudiantes en 5 materias Ciencias Natuales (CNa), Matemáticas (Mat), Francés (Fra), Latín (Lat) y Literatura (Lit)
import pandas as pd
import seaborn as sns
from sklearn.decomposition import PCA
import numpy as np
import matplotlib.pyplot as plt
CNa = [7,5,5,6,7,4,5,5,6,6,6,5,6,8,6,4,6,6,6,7]
Mat = [7,5,6,8,6,4,5,6,5,5,7,5,6,7,7,3,4,6,5,7]
Fra = [5,6,5,5,6,6,5,5,7,6,5,4,6,8,5,4,7,7,4,6]
Lat = [5,6,7,6,7,7,5,5,6,6,6,5,6,8,6,4,8,7,4,7]
Lit = [6,5,5,6,6,6,6,5,6,6,5,4,5,8,6,4,7,7,4,6]
Notas = pd.DataFrame({'CNa':CNa,'Mat':Mat,'Fra':Fra,'Lat':Lat,'Lit':Lit})
Notas
| CNa | Mat | Fra | Lat | Lit | |
|---|---|---|---|---|---|
| 0 | 7 | 7 | 5 | 5 | 6 |
| 1 | 5 | 5 | 6 | 6 | 5 |
| 2 | 5 | 6 | 5 | 7 | 5 |
| 3 | 6 | 8 | 5 | 6 | 6 |
| 4 | 7 | 6 | 6 | 7 | 6 |
| 5 | 4 | 4 | 6 | 7 | 6 |
| 6 | 5 | 5 | 5 | 5 | 6 |
| 7 | 5 | 6 | 5 | 5 | 5 |
| 8 | 6 | 5 | 7 | 6 | 6 |
| 9 | 6 | 5 | 6 | 6 | 6 |
| 10 | 6 | 7 | 5 | 6 | 5 |
| 11 | 5 | 5 | 4 | 5 | 4 |
| 12 | 6 | 6 | 6 | 6 | 5 |
| 13 | 8 | 7 | 8 | 8 | 8 |
| 14 | 6 | 7 | 5 | 6 | 6 |
| 15 | 4 | 3 | 4 | 4 | 4 |
| 16 | 6 | 4 | 7 | 8 | 7 |
| 17 | 6 | 6 | 7 | 7 | 7 |
| 18 | 6 | 5 | 4 | 4 | 4 |
| 19 | 7 | 7 | 6 | 7 | 6 |
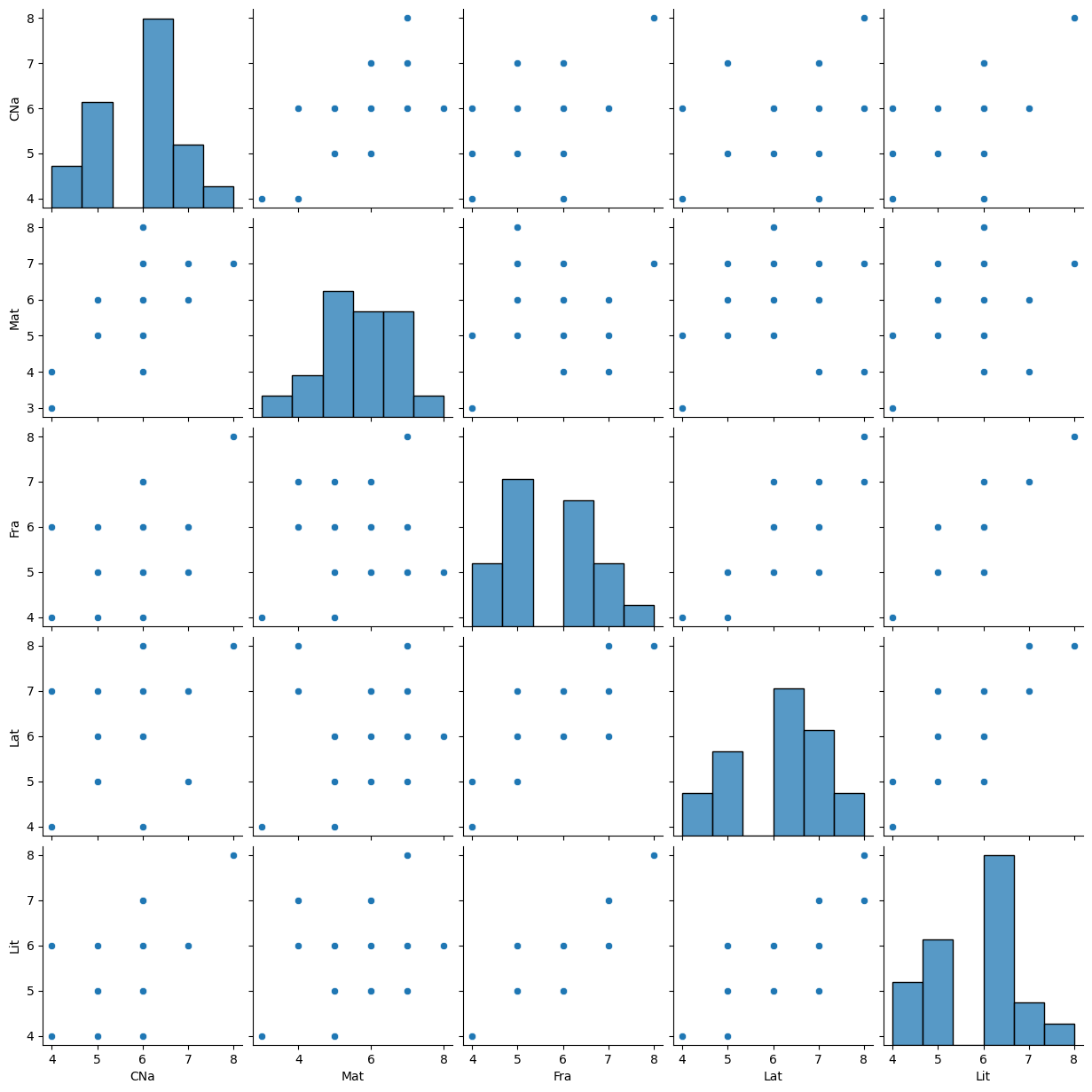
Es pertiente empezar por un análisis explotario para tener una mejor perspectiva de los datos:
Notas.describe()
| CNa | Mat | Fra | Lat | Lit | |
|---|---|---|---|---|---|
| count | 20.000000 | 20.000000 | 20.000000 | 20.000000 | 20.00000 |
| mean | 5.800000 | 5.700000 | 5.600000 | 6.050000 | 5.65000 |
| std | 1.005249 | 1.260743 | 1.095445 | 1.145931 | 1.03999 |
| min | 4.000000 | 3.000000 | 4.000000 | 4.000000 | 4.00000 |
| 25% | 5.000000 | 5.000000 | 5.000000 | 5.000000 | 5.00000 |
| 50% | 6.000000 | 6.000000 | 5.500000 | 6.000000 | 6.00000 |
| 75% | 6.000000 | 7.000000 | 6.000000 | 7.000000 | 6.00000 |
| max | 8.000000 | 8.000000 | 8.000000 | 8.000000 | 8.00000 |
sns.pairplot(Notas)
<seaborn.axisgrid.PairGrid at 0x7f9ba66b6910>
Como habíamos visto, los valores propios corresponden la varianzas explicadas de cada componente y los vectores propios son sus direcciones o pesos (loadings). Es decir:
print(np.corrcoef(Notas.T))
[[1. 0.6561498 0.49706742 0.42034118 0.58398469]
[0.6561498 1. 0.09908375 0.2295101 0.31711567]
[0.49706742 0.09908375 1. 0.81339038 0.84081035]
[0.42034118 0.2295101 0.81339038 1. 0.76622848]
[0.58398469 0.31711567 0.84081035 0.76622848 1. ]]
def fc (x):
val = x-np.mean(x)
return(val)
Notasc = Notas.apply(fc)
from numpy import linalg as LA
from sklearn.decomposition import PCA
from IPython.display import display
S = np.cov(Notas.T) # Matriz de covarianza
e_values, e_vectors = LA.eig(S) # valores y vectores propios
print(e_values)
print(e_vectors)
[3.77855877 1.66135364 0.40960181 0.22023537 0.12498724]
[[-0.39534516 -0.33102922 -0.66155123 0.47595392 0.26446114]
[-0.34882879 -0.79772358 0.37084614 -0.17998543 -0.2683914 ]
[-0.48225721 0.37154122 -0.21520882 -0.01712248 -0.76339838]
[-0.50400573 0.29871462 0.59983776 0.46491466 0.28424781]
[-0.48520809 0.16365646 -0.13675863 -0.72431643 0.44096764]]
Ahora podemos calcular los puntajes de los componetes por individuo:
np.matmul(Notasc,e_vectors) #scores
| 0 | 1 | 2 | 3 | 4 | |
|---|---|---|---|---|---|
| 0 | -0.279154 | -1.913571 | -0.860331 | -0.394234 | 0.282362 |
| 1 | 0.708139 | 0.850534 | 0.242466 | 0.185938 | -0.629896 |
| 2 | 0.337562 | -0.020016 | 1.428359 | 0.487989 | 0.149359 |
| 3 | -0.736643 | -2.081551 | 0.771904 | -0.585259 | 0.033757 |
| 4 | -1.420594 | -0.146877 | -0.246711 | 0.698458 | 0.355851 |
| 5 | 0.463099 | 2.441658 | 0.996251 | -0.369433 | 0.099250 |
| 6 | 1.209194 | 0.343935 | -0.278921 | -0.986171 | 0.290223 |
| 7 | 1.345573 | -0.617445 | 0.228684 | -0.441840 | -0.419136 |
| 8 | -0.654672 | 1.054702 | -0.771052 | -0.079547 | -0.687865 |
| 9 | -0.172414 | 0.683161 | -0.555843 | -0.062425 | 0.075533 |
| 10 | 0.097393 | -1.447484 | 0.537816 | 0.319043 | -0.138819 |
| 11 | 2.661867 | -0.354920 | 0.209805 | 0.479584 | 0.171686 |
| 12 | -0.036035 | -0.278219 | -0.048239 | 0.481906 | -0.633826 |
| 13 | -4.603704 | 0.093480 | -0.641513 | -0.023536 | -0.008693 |
| 14 | -0.387815 | -1.283827 | 0.401058 | -0.405273 | 0.302149 |
| 15 | 4.258876 | 1.272842 | -0.470174 | -0.101313 | 0.159760 |
| 16 | -1.799062 | 2.613512 | -0.078982 | 0.305951 | 0.589989 |
| 17 | -1.992714 | 0.719350 | 0.062873 | -0.518935 | -0.231041 |
| 18 | 2.770528 | -0.984663 | -1.051584 | 0.490624 | 0.151899 |
| 19 | -1.769423 | -0.944601 | 0.124135 | 0.518473 | 0.087459 |
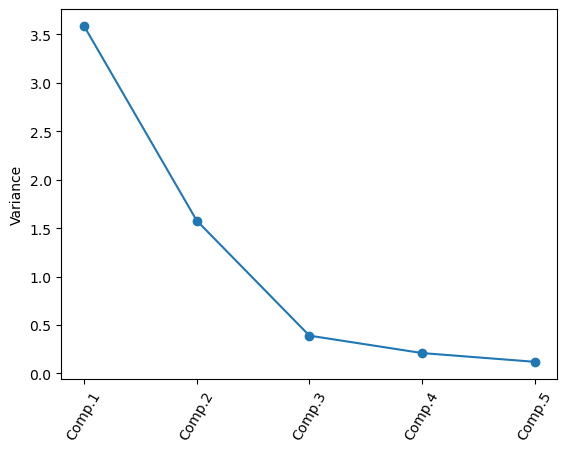
El porcentaje de la varianza explicada por cada componente es:
(e_values/np.sum(e_values))
array([0.60996276, 0.26818793, 0.06612094, 0.03555201, 0.02017636])
Verifiquemos nuestros resultados usando python
pcaMod = PCA(n_components=5).fit(Notas)
pcaMod.explained_variance_ratio_
array([0.60996276, 0.26818793, 0.06612094, 0.03555201, 0.02017636])
pcaMod.components_ # Loadings
array([[ 0.39534516, 0.34882879, 0.48225721, 0.50400573, 0.48520809],
[-0.33102922, -0.79772358, 0.37154122, 0.29871462, 0.16365646],
[-0.66155123, 0.37084614, -0.21520882, 0.59983776, -0.13675863],
[-0.47595392, 0.17998543, 0.01712248, -0.46491466, 0.72431643],
[-0.26446114, 0.2683914 , 0.76339838, -0.28424781, -0.44096764]])
scores = pcaMod.transform(Notas) # scores
scores
array([[ 0.27915411, -1.91357104, -0.86033137, 0.39423402, -0.28236204],
[-0.70813894, 0.85053394, 0.24246638, -0.18593762, 0.62989564],
[-0.33756163, -0.02001625, 1.42835911, -0.48798933, -0.14935915],
[ 0.73664347, -2.08155078, 0.77190377, 0.5852587 , -0.03375732],
[ 1.42059398, -0.146877 , -0.24671081, -0.69845826, -0.35585069],
[-0.46309908, 2.44165783, 0.9962506 , 0.36943263, -0.09925008],
[-1.20919379, 0.34393456, -0.27892119, 0.98617099, -0.29022257],
[-1.34557309, -0.61744548, 0.22868359, 0.44183999, 0.41913648],
[ 0.65467152, 1.05470241, -0.77105231, 0.07954737, 0.68786524],
[ 0.17241431, 0.68316118, -0.55584349, 0.06242489, -0.07553314],
[-0.09739341, -1.44748367, 0.53781626, -0.31904316, 0.13881893],
[-2.66186718, -0.35491959, 0.2098049 , -0.47958435, -0.17168566],
[ 0.03603501, -0.27821886, -0.04823871, -0.48190611, 0.6338259 ],
[ 4.60370426, 0.09348019, -0.64151305, 0.02353642, 0.00869323],
[ 0.38781468, -1.2838272 , 0.40105762, 0.40527327, -0.30214872],
[-4.25887565, 1.27284218, -0.47017392, 0.10131336, -0.15975951],
[ 1.79906227, 2.61351169, -0.07898156, -0.30595095, -0.58998944],
[ 1.99271412, 0.71934991, 0.06287296, 0.51893457, 0.23104118],
[-2.77052775, -0.98466343, -1.0515841 , -0.49062361, -0.15189898],
[ 1.76942278, -0.94460058, 0.12413533, -0.51847283, -0.08745929]])
def pca_summary(pca, standardised_data, out=True):
names = ["PC"+str(i) for i in range(1, len(pca.explained_variance_ratio_)+1)]
a = list(np.std(pca.transform(standardised_data), axis=0))
b = list(pca.explained_variance_ratio_)
c = [np.sum(pca.explained_variance_ratio_[:i]) for i in range(1, len(pca.explained_variance_ratio_)+1)]
columns = pd.MultiIndex.from_tuples([("sdev", "Standard deviation"), ("varprop", "Proportion of Variance"), ("cumprop", "Cumulative Proportion")])
summary = pd.DataFrame(list(zip(a, b, c)), index=names, columns=columns)
if out:
print("Importance of components:")
display(summary)
return summary
sumpca = pca_summary(pcaMod, Notas)
Importance of components:
| sdev | varprop | cumprop | |
|---|---|---|---|
| Standard deviation | Proportion of Variance | Cumulative Proportion | |
| PC1 | 1.894632 | 0.609963 | 0.609963 |
| PC2 | 1.256299 | 0.268188 | 0.878151 |
| PC3 | 0.623796 | 0.066121 | 0.944272 |
| PC4 | 0.457410 | 0.035552 | 0.979824 |
| PC5 | 0.344584 | 0.020176 | 1.000000 |
def screeplot(pca, standardised_values):
y = np.std(pca.transform(standardised_values), axis=0)**2
x = np.arange(len(y)) + 1
plt.plot(x, y, "o-")
plt.xticks(x, ["Comp."+str(i) for i in x], rotation=60)
plt.ylabel("Variance")
plt.show()
screeplot(pcaMod, Notas)
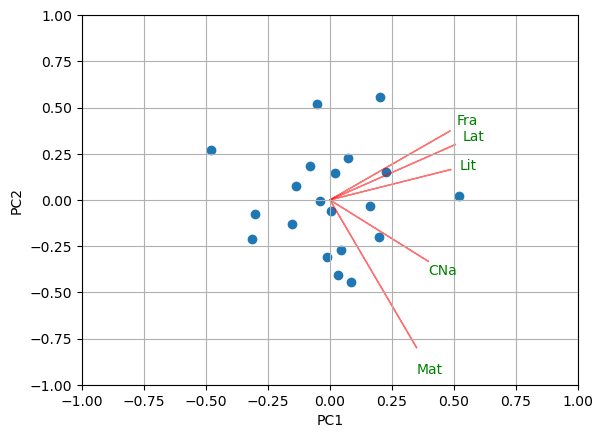
def biplot(score,coeff,labels=None):
xs = score[:,0]
ys = score[:,1]
n = coeff.shape[0]
scalex = 1.0/(xs.max() - xs.min())
scaley = 1.0/(ys.max() - ys.min())
plt.scatter(xs * scalex,ys * scaley)
for i in range(n):
plt.arrow(0, 0, coeff[i,0], coeff[i,1],color = 'r',alpha = 0.5)
if labels is None:
plt.text(coeff[i,0]* 1.15, coeff[i,1] * 1.15, "Var"+str(i+1), color = 'g', ha = 'center', va = 'center')
else:
plt.text(coeff[i,0]* 1.15, coeff[i,1] * 1.15, labels[i], color = 'g', ha = 'center', va = 'center')
plt.xlim(-1,1)
plt.ylim(-1,1)
plt.xlabel("PC{}".format(1))
plt.ylabel("PC{}".format(2))
plt.grid()
biplot(scores[:,0:2],np.transpose(pcaMod.components_[0:2, :]),labels = Notas.columns)
- 1
Teoría obtenida de Peña, D. Análisis de datos multivariantes (2002). Referencias de
FactoMineRvienen de Husson, F. Exploratory multivariate analysis by example using R (2017)
